近年来,结合机器学习算法和多模态脑影像数据的个体行为预测已成为脑发育和脑疾病研究中探究个体水平差异的有力手段。然而许多研究往往以预测精度作为判断其模型质量优劣的重要标准,却在较大程度上忽视了模型的可解释性问题,导致无法精准定位对特定行为表征具有预测性的脑区、环路以及潜在的神经生物学基础。而预测模型的可解释性是理论应用于实践的纽带和桥梁,往往关系到该模型在多大程度上能够辅助临床在个体水平实现脑发育及脑疾病的早期预警、区别诊断和演进判断。
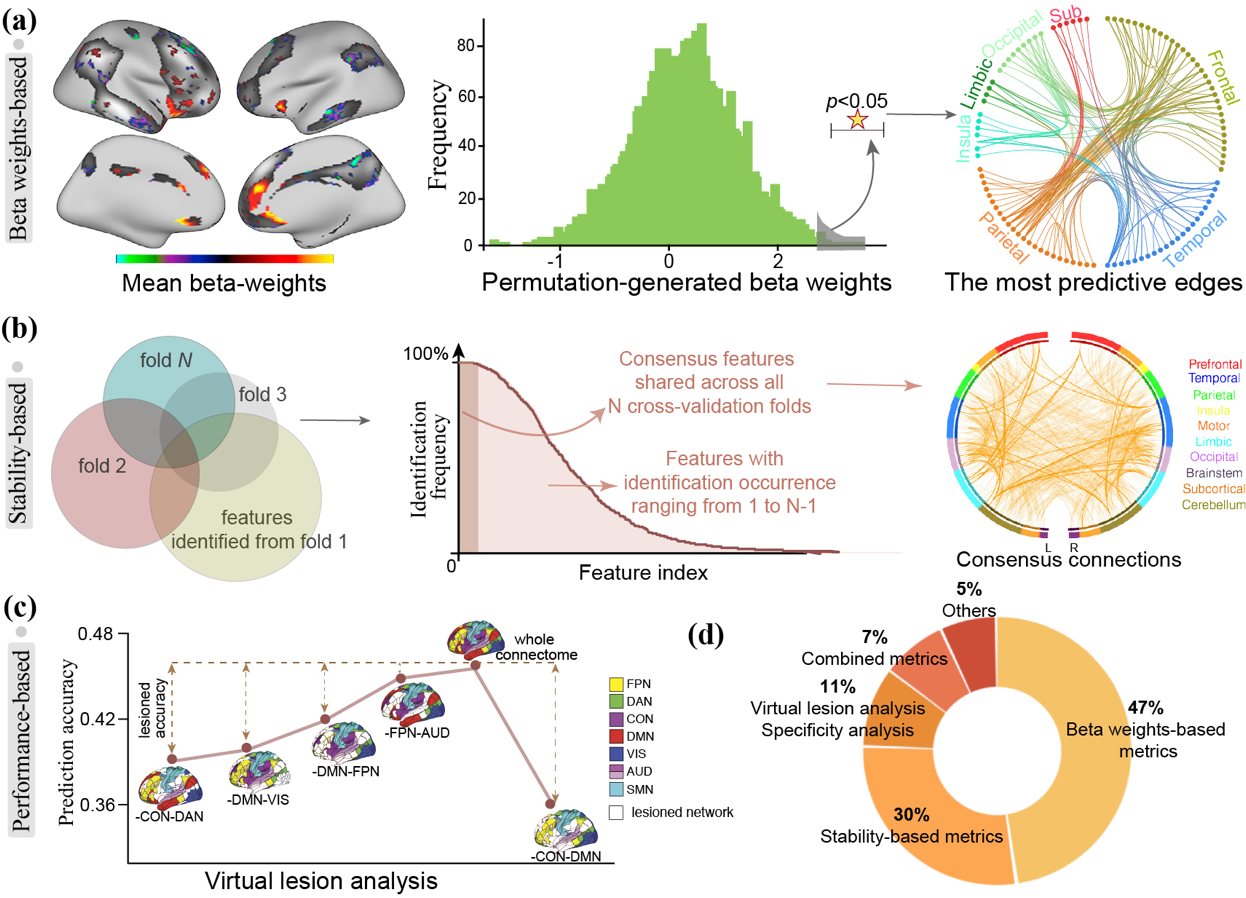
图1. 基于脑影像的个体化预测模型的三种可解释性方法总结
针对上述问题,北京师范大学认知神经科学与学习国家重点实验室隋婧课题组对近十年共326篇相关研究进行了系统分析和提炼,总结归纳了三类预测模型的常用可解释性方法,如图1,并深入探讨了其优缺点及应用场景。
1)基于β系数的方法: 以预测模型的回归系数作为单个特征的贡献大小(图1a),其应用前提是各预测特征进行了归一化处理,并且相互之间不存在多重共线性关系。然而在实际场景中,脑影像特征之间的高度相关性通常会导致不稳定的权重系数。因此,研究者通常使用诸如PCA,LASSO等方法构建或者选择若干不相关的特征,并在此基础上构建模型。
2)基于稳定性的方法: 以特征在多次交叉验证循环或bootstrapping抽样中出现的频次为特征权重度量(图1b)。该方法不受特定模型类型及参数的限制,可用于线性及非线性模型的可解释性分析。实际分析中,研究者通常将在所有交叉验证循环中被反复识别的“一致性特征”作为整体解释相关的脑神经基础。因此该方法适用于从高维影像特征中提取少量特征子集,构建稳定的可泛化模型。
3)基于预测性能的方法:通过排除某一特征来衡量其对模型整体性能的影响,或者基于先验知识利用特定功能网络内的特征进行预测并比较不同网络的预测性能,从而进一步衡量其贡献大小(图1c)。与上述两种方法相比,该方法具有简单直观的优点,可以通过某一功能网络的预测性能直接表示其预测权重,不受模型类型的限制。然而,该方法的使用受研究者先验知识的影响。此外,将全脑功能连接划分到不同网络之中,并在此基础上构建多个预测模型在一定程度上会增加计算负担。
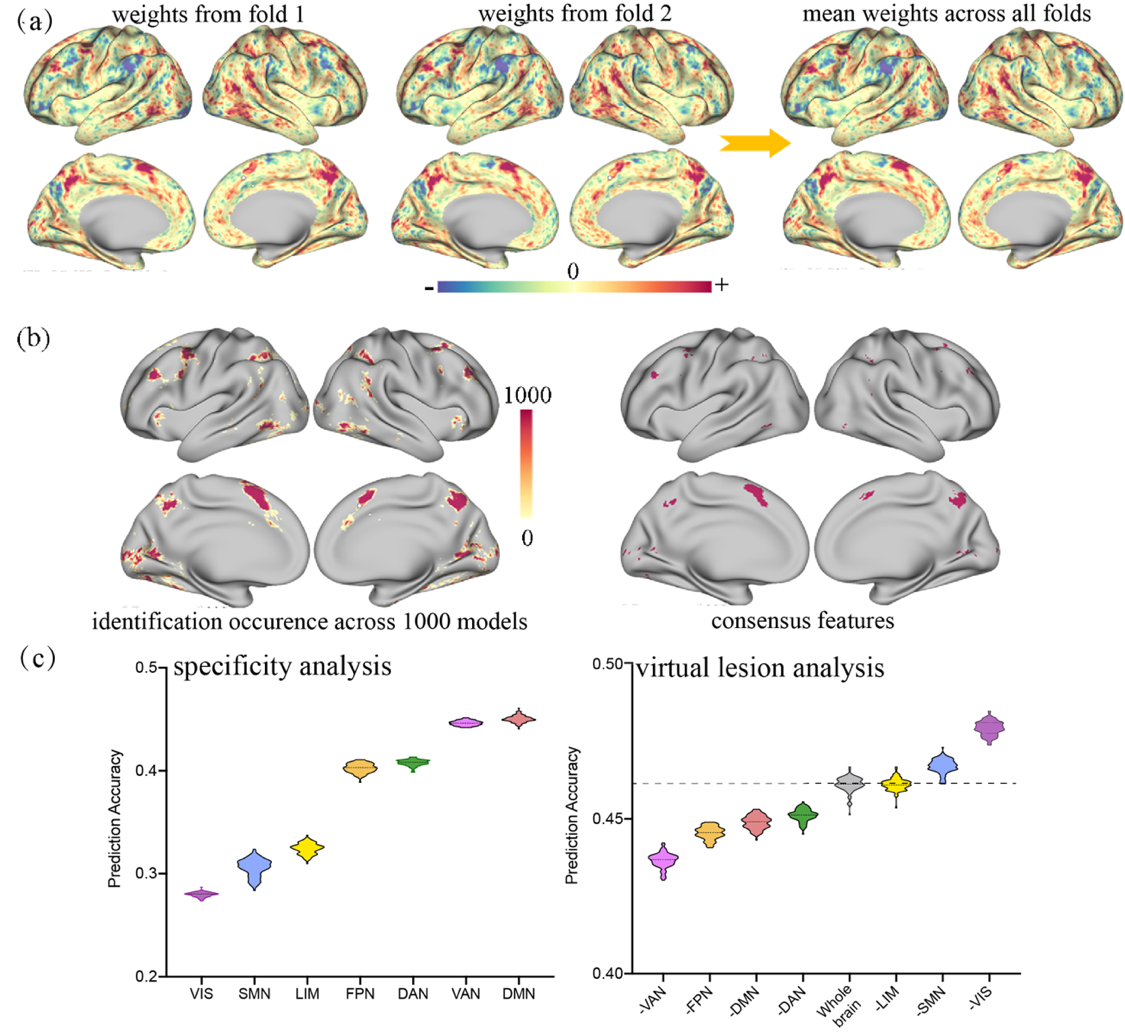
图2. 基于1000例HCP健康被试的功能激活特征对工作记忆能力进行预测,三种模型可解释性方法所识别的预测性特征具有较高的一致性。(a)基于beta系数的可解释性方法:某一交叉验证循环及所有交叉验证循环下平均的Beta系数分布;(b)左图:在重复100次的十折交叉验证循环下,各特征被重复识别的次数分布;右:在1000个模型中均被反复识别的特征;(c)左:基于单一功能网络内的激活特征对工作记忆能力的预测精度分布;右:去掉某一功能网络后,基于剩余功能网络激活特征的预测精度分布。
总体而言,预测模型的可解释性策略选择需要结合各方法的优缺点,综合考虑机器学习算法、样本量、多中心差异等诸多因素,并不存在一种适用所有情况的策略。而当样本量较大时,不同的可解释性方法所识别的特征脑区具有较高的一致性(图2)。此外,为了寻找稳健可靠的影像学标记物,模型得到的预测性脑影像特征的泛化性能检验非常重要,这对于多中心、大规模的数据集研究也提出了新的挑战。
本工作于2022年7月在线发表于信号处理领域权威期刊IEEE Signal Processing Magazine,题为Interpreting Brain Biomarkers: Challenges and solutions in interpreting machine learning-based predictive neuroimaging (https://ieeexplore.ieee.org/document/9810068 )。论文第一作者为姜荣涛博士, 现为耶鲁大学博士后; 通讯作者为北京师范大学认知神经科学与学习国家重点实验室隋婧教授。本研究获得了多项国家自然科学基金的支持。
参考文献
1. J. Sui, R. Jiang, J. Bustillo, et al., “Neuroimaging-based Individualized Prediction of Cognition and Behavior for Mental Disorders and Health: Methods and Promises,” Biol Psychiatry, vol. 88, no. 11, pp. 818-828, 2020.
2. R. A. Poldrack, G. Huckins, and G. Varoquaux, “Establishment of Best Practices for Evidence for Prediction: A Review,” JAMA Psychiatry, vol. 77, no. 5, pp. 534-540, 2020.
3. L. Kohoutova, J. Heo, S. Cha, et al., “Toward a unified framework for interpreting machine-learning models in neuroimaging,” Nat Protoc, vol. 15, no. 4, pp. 1399-1435, 2020.
4. R. Jiang, C. W. Woo, S. Qi, J. Wu and J. Sui, "Interpreting Brain Biomarkers: Challenges and solutions in interpreting machine learning-based predictive neuroimaging," IEEE Signal Processing Magazine, vol. 39, no. 4, pp. 107-118, 2022
5. S. Haufe, F. Meinecke, K. Gorgen, et al., “On the interpretation of weight vectors of linear models in multivariate neuroimaging,” Neuroimage, vol. 87, pp. 96-110, 2014.