2026年1月22日,北京师范大学认知神经科学与学习国家重点实验室赵腾达/贺永团队联合美国宾夕法尼亚大学黄浩教授、北京儿童医院彭芸教授等合作者在Nature Communications在线发表题为 “Hierarchical maturation of structural brain connectomes from birth to childhood” 的研究论文。该研究系统揭示了人脑白质连接网络在儿童早期的层级化时空发育规律及其神经生物学基础。

人脑的神经结构在生命早期历经快速变化,海量突触和轴突连接在多种神经生物因素调控下有序重组,促进跨脑区白质通路的逐步成熟,进而支撑全脑信息的高效整合。然而,人脑白质网络在从出生到儿童早期的时空发育规律及其神经生物基础,目前仍不清楚。为此,该研究整合4组横跨0到8岁的多中心人脑发育影像数据集(416人),结合弥散影像纤维追踪、图论建模、机器学习算法与神经生物注释图谱,系统阐释了人脑白质连接组在多个拓扑维度的层级化时空发育模式及其与多尺度神经生物学特征和微结构的关联。
研究首先基于认知神经科学与学习国家重点实验室与北京大学第三医院、北京儿童医院、美国费城儿童医院合作团队自行采集的两组0-8岁多模态脑影像数据集(共221人,0.15 – 7.9岁),重建了个体水平脑白质连接网络。通过图论分析、中心效应校正与多变量统计,研究发现人脑网络的全局通信效率与抗攻击鲁棒性均随发育显著非线性提升,其快速发育期集中在3.5岁之前(图1)。在节点水平,研究发现脑区通信效率呈现两类截然不同的典型发育轨迹:初级感觉运动皮层缓慢线性增长,高级跨模态联合皮层快速非线性增长,其中外侧额叶、颞叶及岛叶脑区的早期拓扑发育速率(3岁前),可显著预测其在8岁时的通信效率(图2)。随后,研究结合滑动窗与多层网络模型,揭示了前额叶联合皮层独有的两种拓扑重组模式,即通信枢纽重聚集和模块边界重分配(图3)。接下来,研究引入11种人脑神经生物注释图谱进行空间自旋分析,发现脑节点拓扑发育的时空模式与多种神经生物层级模式显著相关,其中通信效率发育速率与感觉运动-联合皮层主轴、皮层髓鞘化轴、皮层进化轴均高度相关,模块灵活度与皮层血流轴、皮层有氧糖酵解轴高度相关(图4)。随后,研究通过提取白质纤维束微结构影像指标,发现联合皮层脑节点早期发育速率对后期拓扑成熟度的预测关系,受其连边纤维的分数各向异性和径向弥散度指标显著中介(图5)。最后,研究在两组公开独立的纵向早期发育脑影像数据集上(195人,0-6岁),验证了结果的可重复性。该研究阐释了从出生到儿童早期人脑白质网络的发育时序、神经生物学约束因素与微结构关联,为探索人脑大尺度结构网络这一复杂智能系统的早期形成规则提供了重要的参照模型和方法支撑。
该论文主要作者包括北京师范大学的赵腾达、寿小婧、贺永,宾夕法尼亚大学的欧阳敏慧、黄浩,北京儿童医院的彭芸,以及北京大学第三医院的袁慧书、刘颖等专家团队。该研究受到国家自然科学基金创新研究群体等项目的资助。
贺永团队主页:http://helab.bnu.edu.cn/
论文链接:https://www.nature.com/articles/s41467-026-68704-w
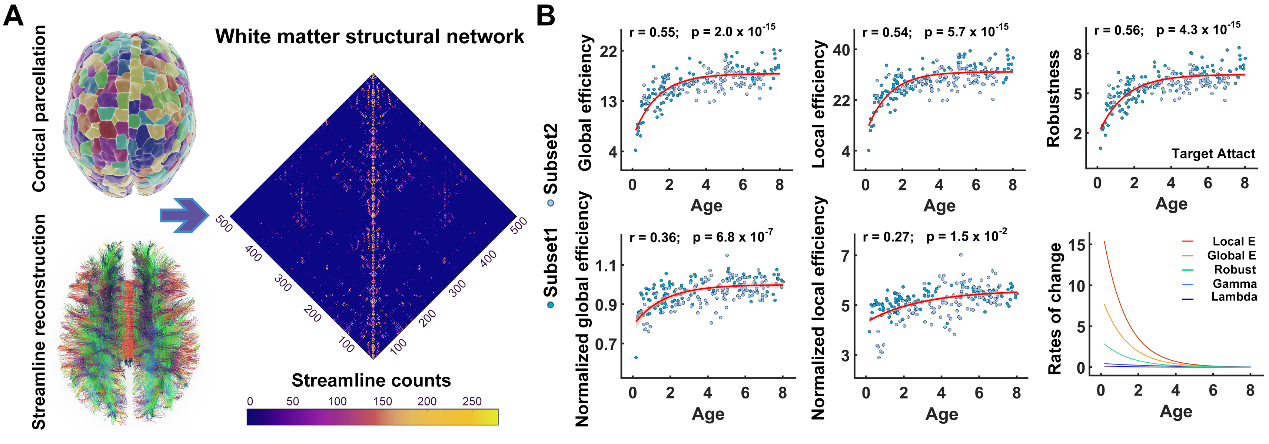
图1:0-8岁脑白质连接组建模及其全局拓扑特征非线性发育模式
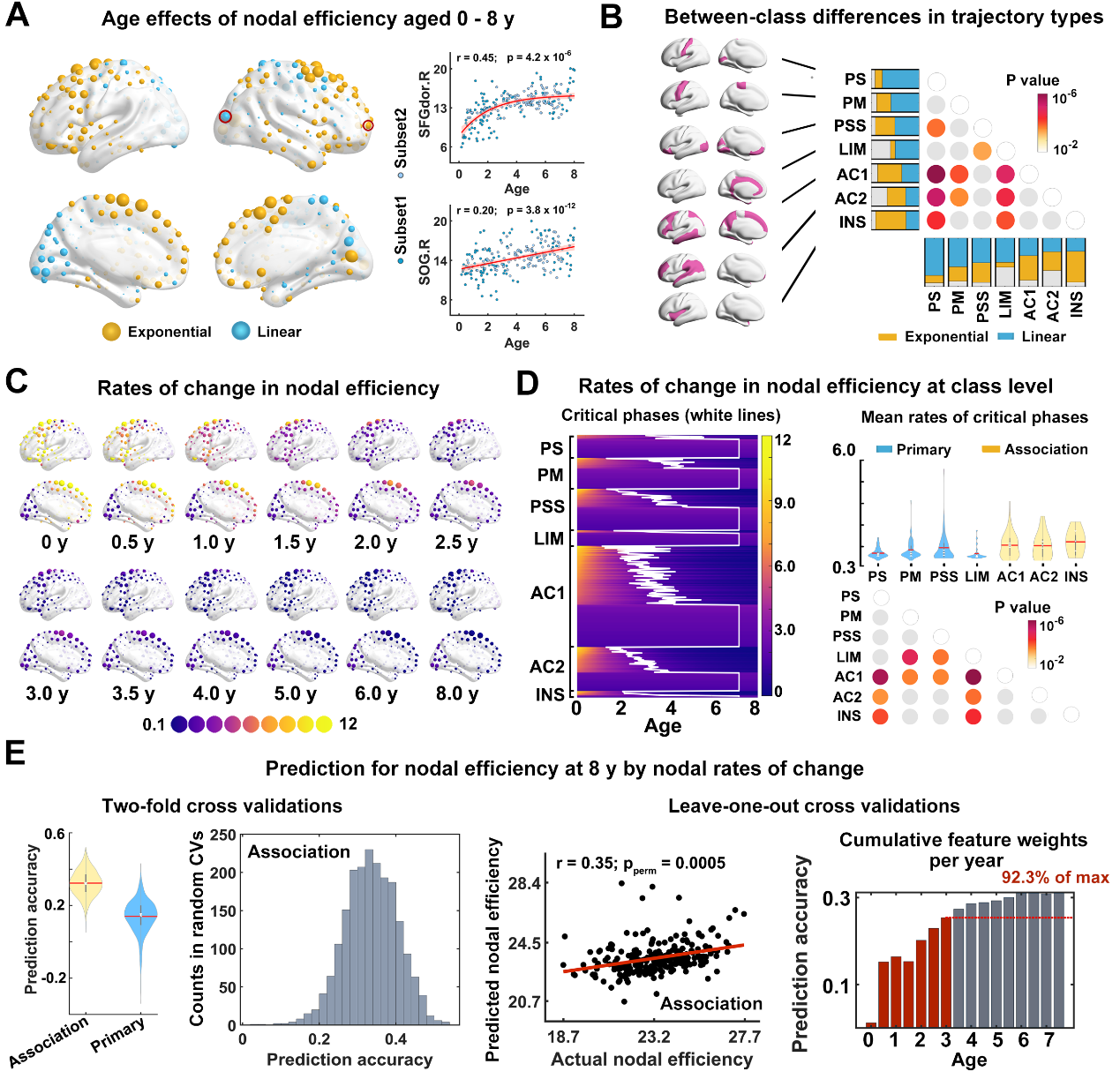
图2:脑节点通信效率层级化时空发育模式及其关键发育期
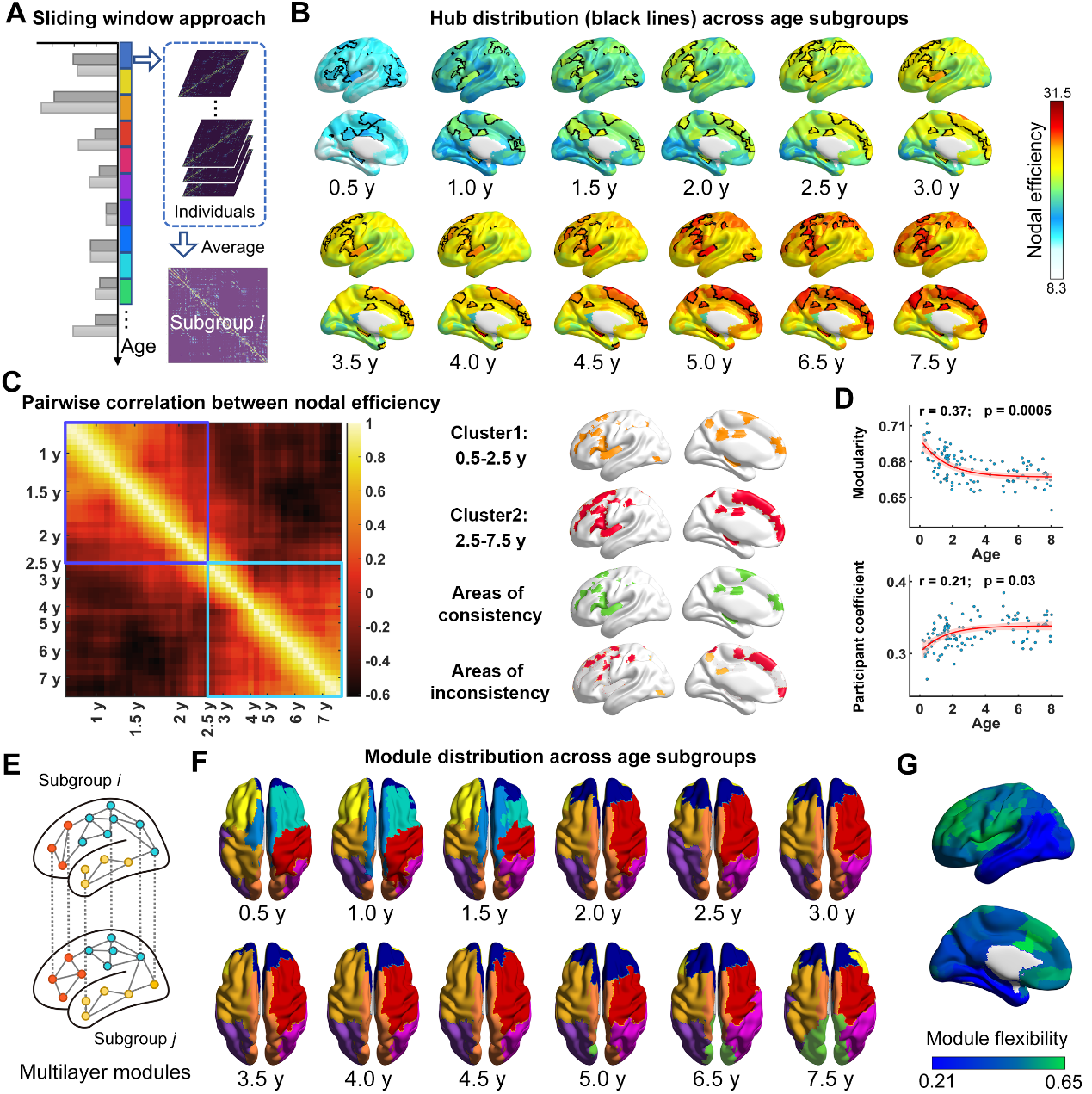
图3:脑白质连接组通信枢纽与模块系统时空发育模式
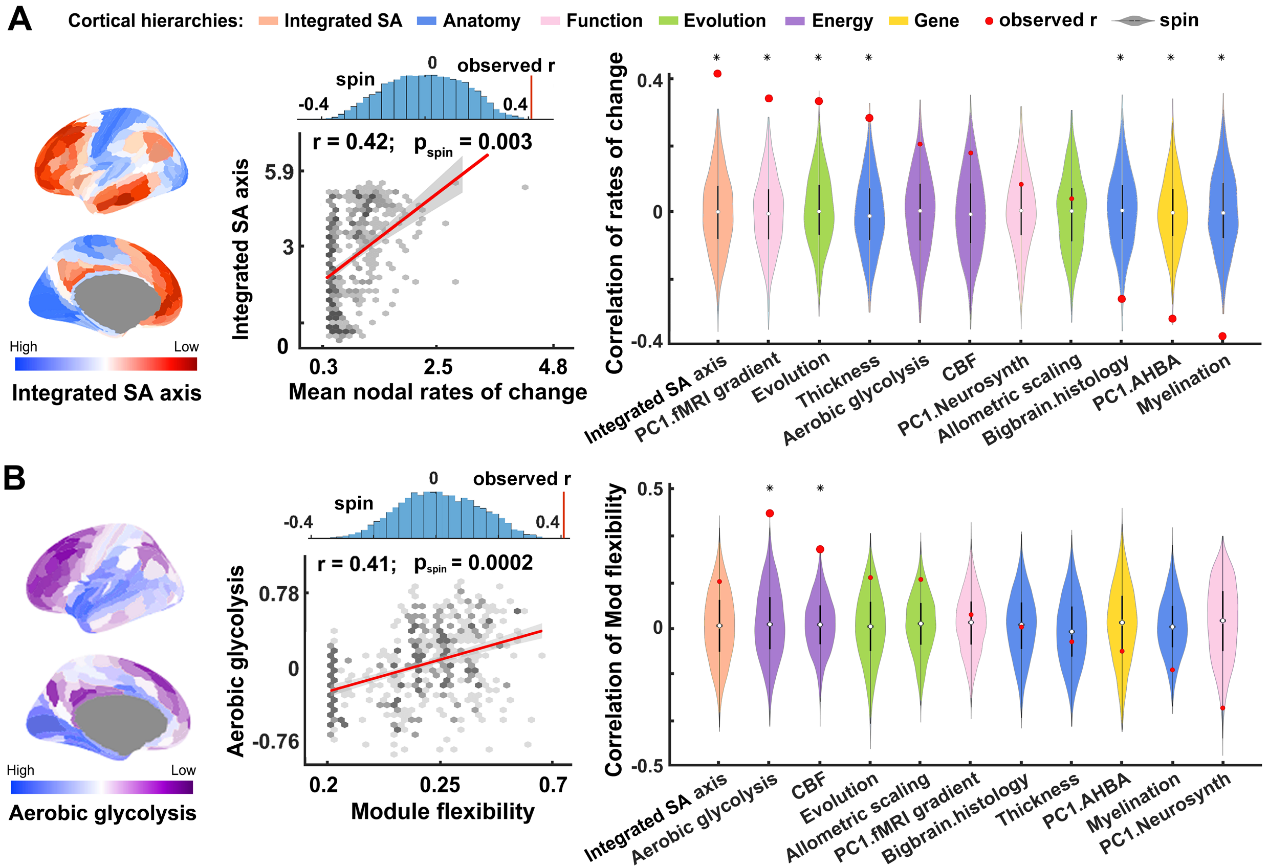
图4:脑网络拓扑发育模式与多尺度神经生物脑轴关联
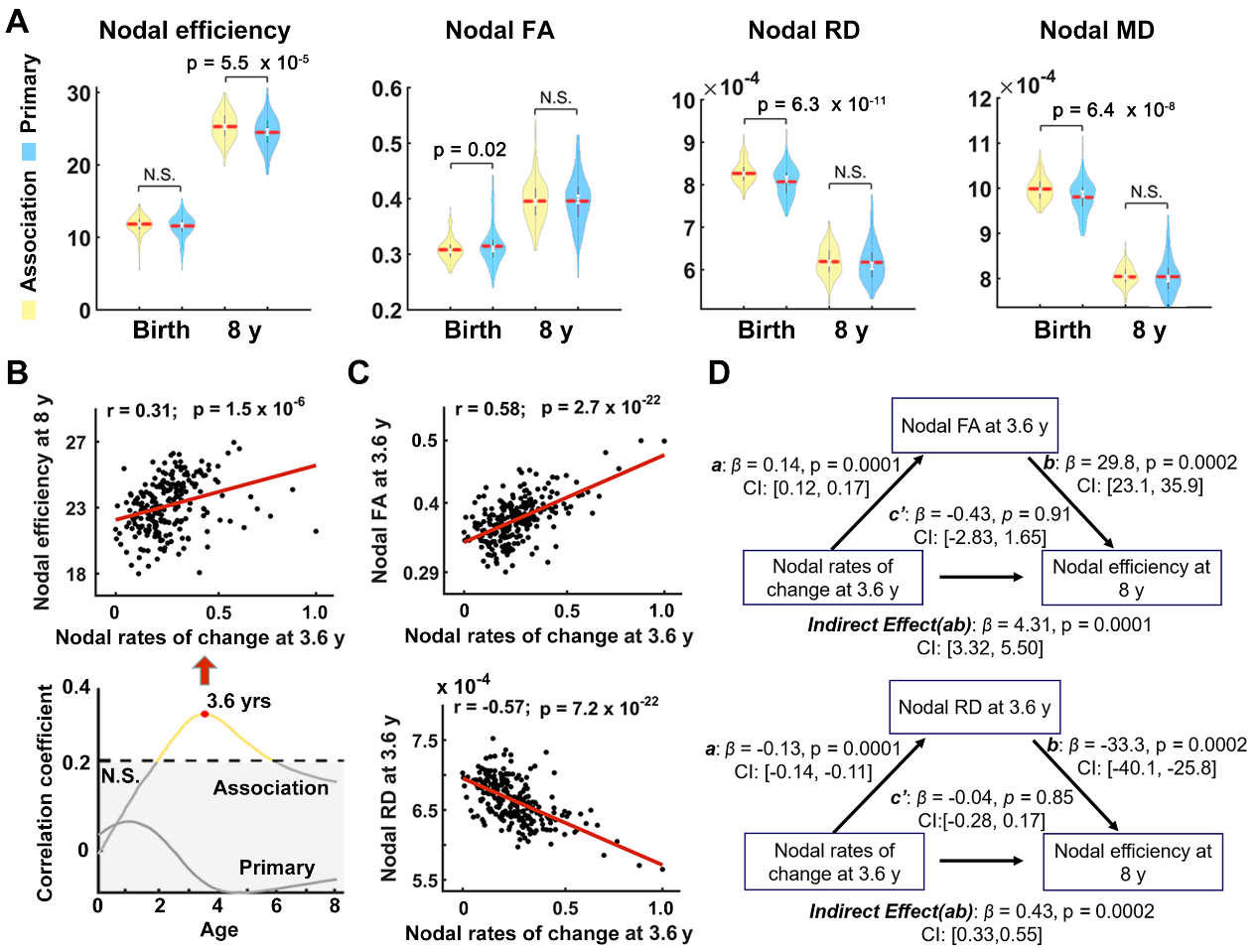
图5:白质微结构中介联合皮层脑区早期拓扑发育速率与8岁通信效率关系
课题组相关文献:
1. Zhao, T., Ouyang, M., Shou, X. J., Zhang, S., Ju, J., Liao, X., Han, M., Sun, L., Wang, X., Xia, Y., Hu, D., Kang, H., Guo, J., Wang, Q., Li, M., Huo, R., Liu, Y., Yuan, H., Peng, Y., … He, Y. (2026). Hierarchical maturation of structural brain connectomes from birth to childhood. Nat Commun. https://doi.org/10.1038/s41467-026-68704-w
2. Liang, X., Sun, L., Xia, M., Zhao, T., Gong, G., Li, Q., Liao, X., Cui, Z., Duan, D., Pang, C., Wang, Q., Yu, Q., Bi, Y., Chen, P., Chen, R., Chen, Y., Chen, T., Cheng, J., Cheng, Y., … He, Y. (2025). Dissecting human cortical similarity networks across the lifespan. Neuron, 113(19), 3275-3295 e3211.
3. Sun, L., Zhao, T., Liang, X., Xia, M., Li, Q., Liao, X., Gong, G., Wang, Q., Pang, C., Yu, Q., Bi, Y., Chen, P., Chen, R., Chen, Y., Chen, T., Cheng, J., Cheng, Y., Cui, Z., Dai, Z.,…He, Y. (2025). Human lifespan changes in the brain's functional connectome. Nat Neurosci, 28(4), 891-901.
4. Liang, X., Sun, L., Liao, X., Lei, T., Xia, M., Duan, D., Zeng, Z., Li, Q., Xu, Z., Men, W., Wang, Y., Tan, S., Gao, J.-H., Qin, S., Tao, S., Dong, Q., Zhao, T., & He, Y. (2024). Structural connectome architecture shapes the maturation of cortical morphology from childhood to adolescence. Nat Commun, 15(1), 784.
5. Zeng, Z., Zhao, T., Sun, L., Zhang, Y., Xia, M., Liao, X., Zhang, J., Shen, D., Wang, L., & He, Y. (2023). 3D-MASNet: 3D mixed-scale asymmetric convolutional segmentation network for 6-month-old infant brain MR images. Hum Brain Mapp, 44(4), 1779-1792.
6. Zhao, T., Xu, Y., & He, Y. (2019). Graph theoretical modeling of baby brain networks. Neuroimage, 185, 711-727.