2023年11月22日,北京师范大学吴倩课题组与钟穗娟课题组在Nature Communications杂志上发表了题为“Single-cell epigenomics and spatiotemporal transcriptomics reveal human cerebellar development”的文章。研究人员联合空间转录组及单细胞转录组、表观遗传组学系统深入地揭示了人类小脑在胚胎发育时期细胞转录组的时空动态变化、细胞类型特点和不同干细胞细胞谱系发育轨迹的时空特征,为深入了解小脑的空间和功能复杂性奠定重要基础。
从灵长类祖先进化到现代人类的过程中,脑容量增加了三倍,认知功能也产生了飞跃式的质变,这取决于几个关键认知脑区体积的增大和功能的进化。认知功能相关的脑区主要包括新皮层、海马体等。团队长期以来关注这些脑区的发育,揭示了认知相关脑区发育的干细胞多样性和细胞命运决定的调控机制,以及这些区域在进化上的异同(Zhong et al.,2018,Zhong et al.,2020,Liu et al.,2017,Zhou et al.,2022,Shi et al.,2021,Zeng et al.,2023,Fan et al.,2018)。科学家们前期对认知功能相关脑区的研究主要集中在了大脑皮层及海马体中,但最近也有研究指出小脑不仅在机体运动功能上发挥着重要的作用,也能参与到高级认知功能。因此,了解小脑的发育与进化机制,是我们解析小脑高级认知功能的形成基础必不可少的一步。
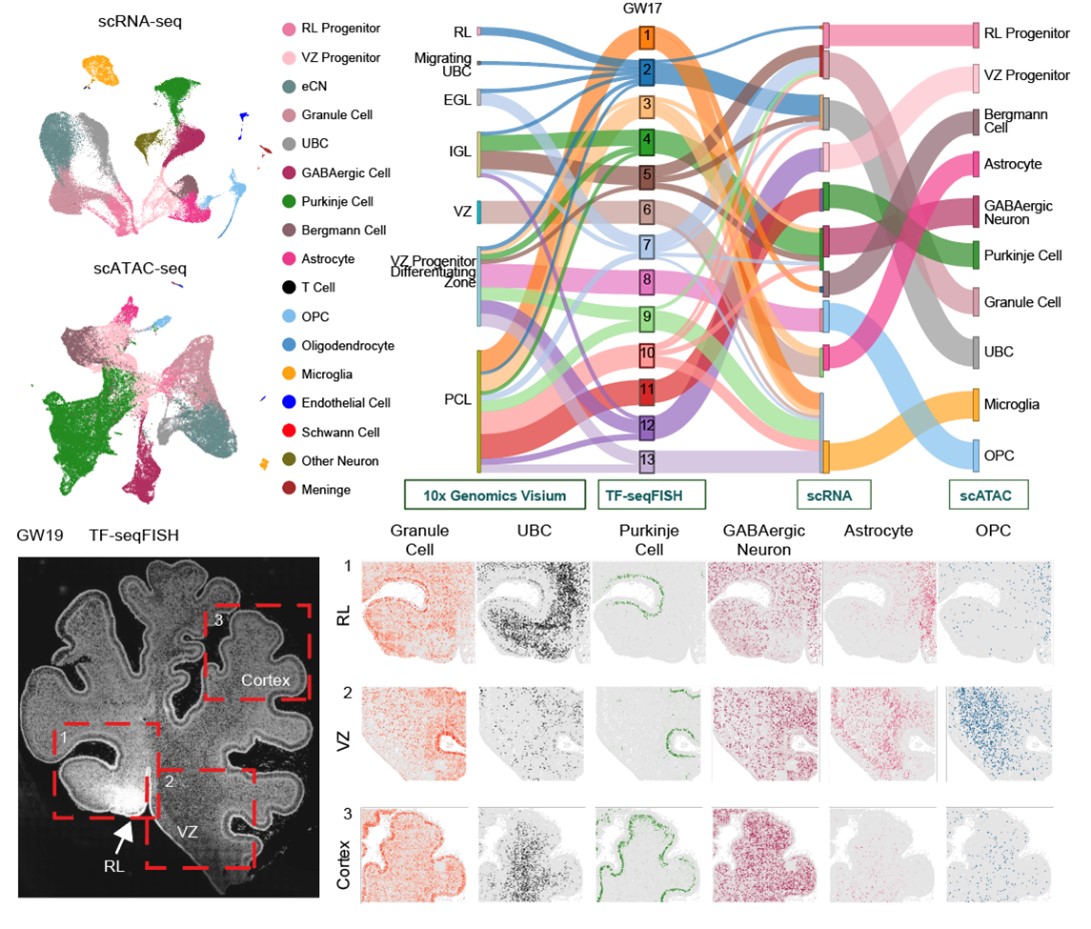
图1. 多组学联合揭示人类小脑动态发育特征
基于前期的工作积累,研究人员为了更好的描绘和揭示小脑发育的时空特征,经过多年攻关,研发出了基于单分子成像的单细胞空间转录组技术TF-seqFISH(Transcription Factor sequential Fluorescence In Situ Hybridization),该技术可以检测人1085个转录因子的mRNA在单个细胞中的亚细胞定位和表达量。利用单细胞空间转录组、单细胞转录组和单细胞表观遗传组学数据联合分析,系统描绘了人小脑胚胎发育的细胞和分子空间动态景观,并鉴定细胞亚群和基因表达特征(图1)。通过细胞聚类和发育轨迹分析,阐明小脑神经干细胞类型的多样性,主要体现在分子表达、空间分布、成熟状态和分化潜能等方面的差异。在该研究中,我们揭示了维持细胞命运层级性分化的重要调控因子,构建了不同神经干细胞亚型向不同终端细胞(包括多种神经元和胶质细胞)分化过程中的基因调控网络,表明不同细胞类型的命运特征决定发生于发育早期,并受到一系列不同的关键转录因子的组合调控(图2)。
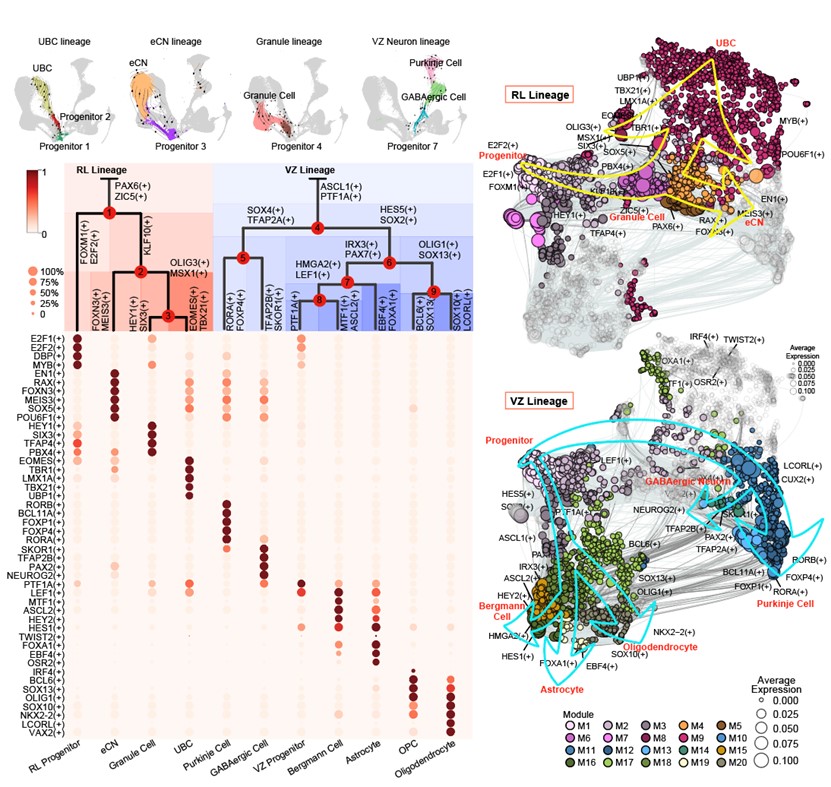
图2.多层级转录因子调控决定了细胞分化命运和发育路径
研究人员进一步的研究揭示了人类小脑浦肯野细胞在增殖分化过程中的重要调控因子PTF1A及RORA,RORB,PCP4等关键转录因子的调控作用,提出不同的浦肯野细胞在分化时间和空间上有独特的表型及分子特征。RORB作为浦肯野细胞的新亚型重要标记性蛋白,从浦肯野细胞早期就参与浦肯野细胞的分化调控,影响浦肯野细胞的递质投送,突触发生等突触形成期间关键的功能运作。颗粒细胞作为小脑中数量最多的细胞类型,探究该类细胞的时空发育特征一直是揭秘小脑必不可少的关键要素。研究人员发现,颗粒细胞的增殖和分化贯穿整个小脑胚胎发育期,并在早期通过维持NOTCH信号通路促使干细胞转向分化为外颗粒细胞,从小脑干细胞区域菱唇(Rhombic Lip)向外颗粒层进行迁移,并在外颗粒层保持极高的增殖活性。而在晚期则激活WNT信号通路,HEY1作为小脑胚胎发育期外颗粒细胞的重要调控分子,能够驱使外颗粒细胞迁移到内颗粒层,并开始进行突触发生,构建神经网络。值得一提的是,研究人员发现颗粒细胞在不同的小脑小叶中具有独特的空间分子特征(图3)。
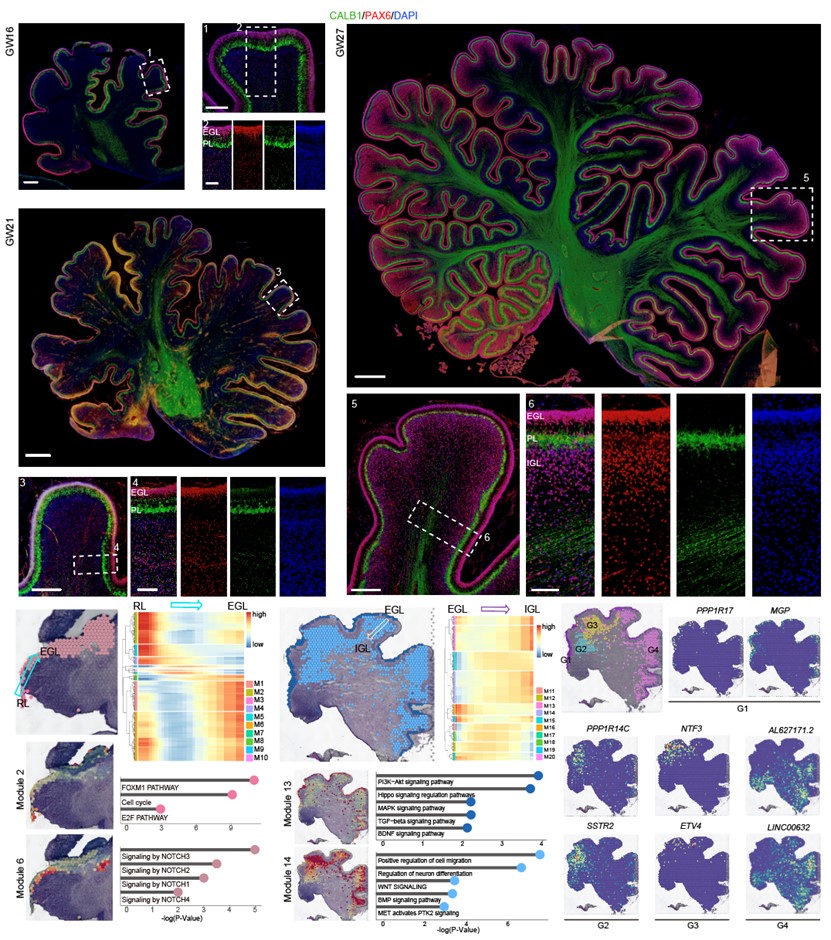
图3.调控颗粒细胞发育和迁移的机制
通过跨物种比较分析,研究人员发现与小鼠相比,人类小脑具有独特的神经细胞亚型并展示着人类特有的分子特征。有意思的是,人类小脑的颗粒细胞之所以具有超乎想象的增殖分化能力与人类特异性基因ARHGAP11B的表达密不可分,研究人员在小鼠胚胎发育早期促使小鼠小脑外源表达ARHGAP11B蛋白,能够有效地增大小脑的周长与横截面积,驱使颗粒细胞的大量增殖分化,并形成了更多的沟回折叠,表明ARHGAP11B在人类小脑进化中起了重要作用。最后,研究人员揭示了单核苷酸多态性(SNPs)与小脑疾病的关系,展示了基因编码区和非编码区发生单位点突变,同样能够对小脑发育疾病的产生有着重要的影响。总之,该研究提供了胚胎发育期人类小脑发育的细胞图谱和基因表达特征,为深入了解其空间和功能复杂性、发育异常引起的疾病机理奠定了重要基础。
本研究得到国家自然科学基金、国家重点研发计划、新基石研究员等项目的资助,以及王晓群教授的大力支持。北京师范大学钟穗娟副研究员、生物物理所博士生王梦迪、黄露苇、北京师范大学博士生陈优巧为论文共同第一作者。北京师范大学吴倩教授、钟穗娟副研究员为论文的共同通讯作者。
论文链接:https://www.nature.com/articles/s41467-023-43568-6
参考文献:
1. Zhong, S., Zhang, S., Fan, X., Wu, Q., Yan, L., Dong, J., Zhang, H., Li, L., Sun, L., Pan, N., et al. (2018). A single-cell RNA-seq survey of the developmental landscape of the human prefrontal cortex. Nature 555, 524-528. 10.1038/nature25980.
2. Zhong, S., Ding, W., Sun, L., Lu, Y., Dong, H., Fan, X., Liu, Z., Chen, R., Zhang, S., Ma, Q., et al. (2020). Decoding the development of the human hippocampus. Nature 577, 531-536. 10.1038/s41586-019-1917-5.
3. Liu, J., Liu, W., Yang, L., Wu, Q., Zhang, H., Fang, A., Li, L., Xu, X., Sun, L., Zhang, J., et al. (2017). The Primate-Specific Gene TMEM14B Marks Outer Radial Glia Cells and Promotes Cortical Expansion and Folding. Cell Stem Cell 21, 635-649 e638. 10.1016/j.stem.2017.08.013.
4. Zhou, X., Lu, Y., Zhao, F., Dong, J., Ma, W., Zhong, S., Wang, M., Wang, B., Zhao, Y., Shi, Y., et al. (2022). Deciphering the spatial-temporal transcriptional landscape of human hypothalamus development. Cell Stem Cell 29, 328-343 e325. 10.1016/j.stem.2021.11.009.
5. Shi, Y., Wang, M., Mi, D., Lu, T., Wang, B., Dong, H., Zhong, S., Chen, Y., Sun, L., Zhou, X., et al. (2021). Mouse and human share conserved transcriptional programs for interneuron development. Science 374, eabj6641. 10.1126/science.abj6641.
6. Zeng, B., Liu, Z., Lu, Y., Zhong, S., Qin, S., Huang, L., Zeng, Y., Li, Z., Dong, H., Shi, Y., et al. (2023). The single-cell and spatial transcriptional landscape of human gastrulation and early brain development. Cell Stem Cell 30, 851-866 e857. 10.1016/j.stem.2023.04.016.
7. Fan, X., Dong, J., Zhong, S., Wei, Y., Wu, Q., Yan, L., Yong, J., Sun, L., Wang, X., Zhao, Y., et al. (2018). Spatial transcriptomic survey of human embryonic cerebral cortex by single-cell RNA-seq analysis. Cell Res 28, 730-745. 10.1038/s41422-018-0053-3.